El largo plazo Programa Folding @ Home El crowdsourcing de la enormemente compleja tarea de resolver las interacciones moleculares ha alcanzado un hito importante ya que miles de nuevos usuarios se registran para poner sus computadoras a trabajar. La red ahora comprende un "exaflop" de potencia informática: 1,000,000,000,000,000,000 operaciones por segundo.
Folding @ Home comenzó hace unos 20 años como una forma, luego novedosa, y pionera de la ahora hibernante SETI @ Home, para romper los problemas de computación y distribuirlos a las personas para su ejecución. Equivale a una supercomputadora cruda distribuida por todo el mundo, y aunque no es tan efectiva como una supercomputadora "real" en la voladura a través de cálculos, puede hacer un trabajo corto de problemas complejos.
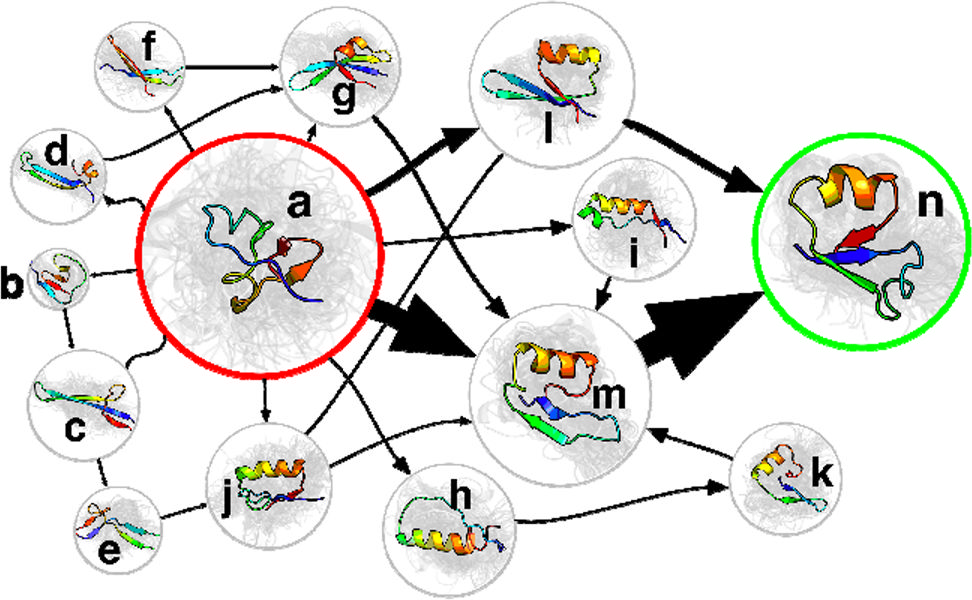
El problema en cuestión que aborda esta herramienta (administrado por un grupo de la Universidad de Washington en St. Louis) es el del plegamiento de proteínas. Las proteínas son una de las muchas estructuras químicas que hacen que nuestra biología funcione, y van desde moléculas pequeñas y relativamente bien entendidas hasta moléculas realmente enormes.
Lo que pasa con las proteínas es que cambian de forma según las condiciones: temperatura, pH, presencia o ausencia de otras moléculas. Este cambio de forma es a menudo lo que los hace útiles, por ejemplo, una proteína de kinesina cambia de forma como un par de patas que toman medidas para llevar una carga útil a través de una célula. Otra proteína como un canal iónico se abrirá para dejar pasar a los átomos cargados solo si hay otra proteína presente, que encaja en ella como una llave en una cerradura.
Algunos de estos cambios, o convoluciones, están bien documentados, pero la mayoría son totalmente desconocidos. Pero a través de una simulación robusta de las moléculas y sus alrededores, podemos descubrir nueva información sobre las proteínas que pueden conducir a descubrimientos importantes. Por ejemplo, ¿qué pasaría si pudieras demostrar que una vez que el canal iónico está abierto, otra proteína podría bloquearlo de esa manera por más tiempo de lo normal o cerrarlo rápidamente? Encontrar ese tipo de oportunidades es de lo que se trata este tipo de ciencia molecular.
Lamentablemente también es extremadamente Cálculo costoso. Estas interacciones intermoleculares e intramoleculares son el tipo de cosas que las supercomputadoras pueden destruir sin parar para cubrir todas las posibilidades. Hace veinte años, las supercomputadoras eran mucho más raras de lo que son hoy, por lo que Folding @ Home comenzó como una forma de hacer este tipo de carga de computación pesada sin comprar una configuración Cray de $ 500 millones.
El programa ha estado funcionando todo el tiempo, y probablemente recibió un impulso cuando SETI @ Home lo recomendó como una alternativa a sus muchos usuarios. Pero la crisis del coronavirus ha hecho que la idea de contribuir con los recursos de uno a una causa mayor sea muy atractiva y, como tal, ha habido un gran aumento de usuarios, tanto que los servidores están luchando para resolver los problemas de las computadoras de todos.
El hito que está celebrando es el logro de un exaflop de poder de procesamiento, que creo que es un sextillón (mil millones de billones) de operaciones por segundo. Una operación es una operación lógica, como AND o NOR, y varias de ellas juntas forman expresiones matemáticas, que eventualmente se suman a cosas útiles como decir "a temperaturas superiores a 38 grados Celsius, esta proteína se deforma para permitir que un medicamento se una en este sitio y deshabilítelo ".
La computación Exascale es el próximo objetivo de las supercomputadoras; Intel y Cray están construyendo computadoras a gran escala para los Laboratorios Nacionales que se espera que entren en línea en los próximos años, pero las supercomputadoras más rápidas disponibles en la actualidad operan a una escala de cientos de petaflops, o aproximadamente la mitad a un tercio de la velocidad como un exaflop.
Naturalmente, estas dos cosas no son directamente comparables: Folding @ Home está organizando el valor de una potencia de cálculo de un exaflop, pero no funciona como una sola unidad que trabaja en un solo problema, como los sistemas exascale están construidos para. El exacto está ahí para dar un sentido de escala.
¿Este tipo de análisis conducirá a tratamientos con coronavirus? Quizás más tarde, pero casi seguro que no en el futuro inmediato. La proteómica es "investigación básica" en el sentido de que se trata de comprender mejor el mundo que nos rodea (y dentro de nosotros).
COVID-19 (como Parkinson, Alzheimer, ALS y otros) no es un problema único, sino un conjunto de incógnitas grandes y mal delimitadas; su proteoma y las interacciones relacionadas son parte de ese conjunto. El punto no es tropezar con una bala mágica, sino sentar las bases para la comprensión de modo que cuando evaluamos posibles soluciones, podamos elegir la correcta incluso un 1% más rápido porque sabemos que esta molécula en ese la situación actúa como entonces.
Como señaló el proyecto en una publicación de blog que anuncia el lanzamiento del trabajo relacionado con el coronavirus:
Esta ola inicial de proyectos se enfoca en comprender mejor cómo estos coronavirus interactúan con el receptor ACE2 humano requerido para la entrada viral en las células huésped humanas, y cómo los investigadores podrían interferir con ellos a través del diseño de nuevos anticuerpos terapéuticos o moléculas pequeñas que podrían alterar su interacción
Si quieres ayudar, puedes descargue el cliente Folding @ Home y done sus ciclos de CPU y GPU de repuesto a la causa.